上海科技大学信息科学与技术学院郑杰课题组近日提出了基于知识图谱和可解释图神经网络的新型人工智能模型KR4SL,在预测并解释基于合成致死机制的抗癌药物靶点方面取得进展。该成果被生物信息学旗舰会议——第31届分子生物学智能系统会议和第22届欧洲计算生物学会议(the 31stAnnual International Conference on Intelligent Systems for Molecular Biology and the 22nd Annual European Conference on Computational Biology, ISMB/ECCB 2023) 以会议论文形式接收。该工作同时被收录在生物信息学领域核心期刊《生物信息学》(Bioinformatics)的ISMB/ECCB 2023会议特刊。郑杰课题组博士生张可在大会上做口头报告。ISMB/ECCB 2023大会共收到336篇投稿论文,仅有60篇被选为会议论文,接收率为17.9%。
合成致死 (synthetic lethality,SL)是基因之间的一种遗传相互作用,即单独一个基因失活不影响细胞活力,而抑制两种基因则导致细胞死亡。利用SL可以选择性地杀死癌细胞,即使某些突变基因无法直接成为药物靶标,也可以针对与之有SL关系的伙伴基因来攻击癌细胞,而不损害正常细胞。当前识别SL基因对的方法包括实验筛选和计算预测,实验筛选成本高昂且可能有脱靶效应。同时,大部分已识别的SL基因的生物机制仍然不清晰。知识图谱中包含大量的结构化生物医药知识,为使用人工智能技术预测和解释SL基因对提供支持。然而,目前的计算方法主要依赖于从知识图谱中提取子图来学习基因的表示,无法充分利用知识图谱中的所有结构信息,同时也缺乏结果的可解释性。
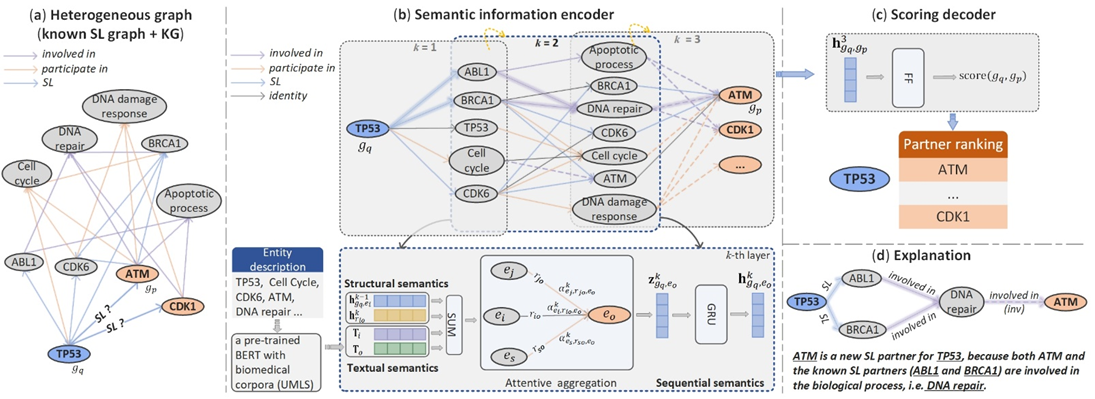
图:KR4SL模型。(a) 整合已有的SL关系和知识图谱构建新的异构图。(b)上图表示为给定基因搜索关系有向图并找到可能的候选SL伙伴基因。下图表示整合三种信息并利用注意力机制聚合消息。(c)根据基因对的表示为候选基因打分。(d)关系路径解释预测过程和潜在合成致死机制。
针对现有人工智能方法在知识图谱利用方面的不足,研究人员提出了一个基于知识图推理(knowledge graph reasoning)的可解释图神经网络模型KR4SL。KR4SL基于动态规划思想高效地为多个基因对寻找关系有向图(relational directed graph),同时整合关系有向图的结构信息、关系路径的序列信息和实体的文本信息,进而充分学习基因对的语义表示,以预测新的SL关系。最为突出的是,KR4SL还实现了模型的可解释性。它通过加权的关系路径进行推理,使得用户可以清晰地追踪整个预测过程,了解预测的原因和逻辑。大量的实验结果表明KR4SL预测能力优于最新的基准方法,并且在两种实际场景下表现出了强大的泛化能力。
该成果主要由上海科技大学信息科学与技术学院郑杰课题组完成,并以“KR4SL: knowledge graph reasoning for explainable prediction of synthetic lethality”为题发表。信息科学与技术学院2019级博士生张可为论文第一作者,郑杰教授为通讯作者。其他作者包括新加坡南洋理工大学资深研究科学家刘勇、新加坡科技研究局资深科学家吴敏和郑杰课题组博士生冯艺苗。
文章链接:https://academic.oup.com/bioinformatics/article/39/Supplement_1/i158/7210467




 沪公网安备 31011502006855号
沪公网安备 31011502006855号